2015年3月10日,大阳城国际娱乐官网自动化系、清华信息国家实验室生物信息学研究部、合成与系统生物学中心汪小我和谢震研究组在《美国科学院院刊》PNAS在线发表题为《模型指导下利用合成基因线路对microRNA介导的竞争性内源RNA调控进行定量分析》(Model-guided quantitative analysis of microRNA-mediated regulation on competing endogenous RNAs using a synthetic gene circuit)的研究论文。该文系统分析了影响microRNA介导的竞争性内源RNA效应(competing endogenous RNA, ceRNA),发现了若干影响microRNA定量调控规律的因素,并提出了RNA干涉技术(RNAi)的改进原则。
MicroRNA是一类短的内源性RNA,通过与靶RNA的结合来抑制靶基因的表达,在动植物中起到重要的转录后调控作用。每一类microRNA可以同时结合多种类型的靶RNA。当microRNA的表达量一定时,各种靶RNA会与microRNA竞争性结合,从而影响到microRNA对其他靶基因的调控,这一现象被称为竞争性内源RNA,即ceRNA效应。与此同时,困扰目前广泛使用的RNA干扰技术(RNAi)的脱靶现象也与该效应密切相关。如何系统地、定量地刻画ceRNA效应对理解microRNA调控网络和改进RNA干扰技术都具有重要意义。
CeRNA效应研究的一个难点在于细胞内microRNA调控系统由复杂的多对多非线性调控关系组成,难以仅对其中一个局部进行扰动和观察。项目组采用了将系统生物学建模分析与合成生物学实验相互结合的策略来研究这一复杂系统。研究人员建立了用于刻画microRNA调控的数学模型,并利用合成生物学的手段构建合成基因线路并植入人类细胞中以模拟ceRNA调控关系。通过模型预测指导实验验证,并利用实验结果修正模型和拟合模型参数。论文证实了靶RNA和microRNA浓度对ceRNA效应的阈值现象,报道了microRNA与靶位点的结合能力对ceRNA效应强度影响的函数关系,阐述了microRNA通路和RNAi通路竞争效应的不对称性,并从理论上提出了RNA干扰技术的改进方向。同时,该工作表明将计算系统生物学理论分析与合成生物实验相结合是解析基因复杂调控规律的一种有力方式。
该项目得到了国家重点基础研究发展计划(973)、国家自然科学基金优秀青年基金和清华信息国家实验室的资助。
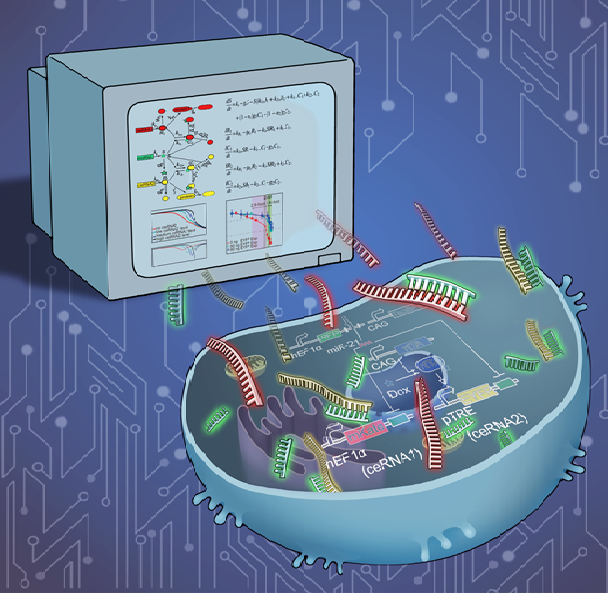
将系统生物学建模分析与合成生物学实验相结合解析microRNA定量调控规律

